Procesado de imágenes DICOM
Hemos realizado el proyecto en Python por el conocimiento generado en anteriores entregas de la asignatura. Python ofrece una orientación a objetos beneficiosa para programar de forma modular y permite el acceso a librerías de gestión para el trabajo que nos ocupa (General Python FAQ - Python 3.14.0., s.f.).
Específicamente, se han utilizado las librerías siguientes:
matplotlib, para generar Interfaces de Usuario Gráficas (GUI) con el uso del kit de herramientas widgets. Para el cuadro de diálogo inicial se utiliza tkinter.filedialog (Tkinter - Python Interface to Tcl/Tk, s.f.) (Matplotlib Documentation - Matplotlib 3.10.8 Documentation, s.f.)pydicom, como marco para leer y decodificar imágenes en estándar DICOMnumpy. La librería numpy permite la gestión y análisis de objetos numéricos. Utilizaremos su capacidad para trabajar con arrays o matrices (Numpy Documentation, s.f.)os, para poder trabajar con llamadas a sistema y rutas de acceso a disco.Podemos resumir la funcionalidad de la aplicación de la siguiente forma:
Slider.flatten de NumPy, para pasar los datos a una dimensión.Vistas las características de la aplicación, procedemos a explicar el código del programa:
Al trabajar en un lenguaje interpretado como Python, es preciso que las declaraciones de clases, métodos y funciones se hagan antes de su llamada. Se considera innecesaria la documentación de la aplicación, existiendo para ello comentarios y bloques de documentación en el fichero fuente.
Considerando que la aplicación funciona únicamente en sistemas UNIX, se adjuntan las siguientes capturas de pantalla en una instalación de Debian 13 con arquitectura de 64 bits:
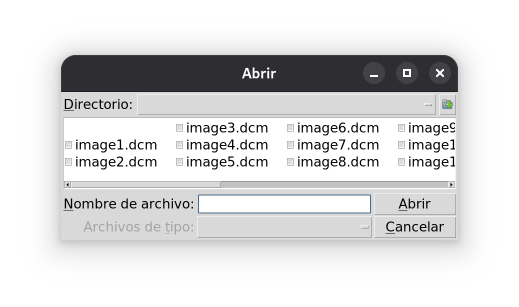
La primera pantalla que aparece es el selector de archivo:

Después la imagen se procesa, llegando a la pantalla principal del programa:
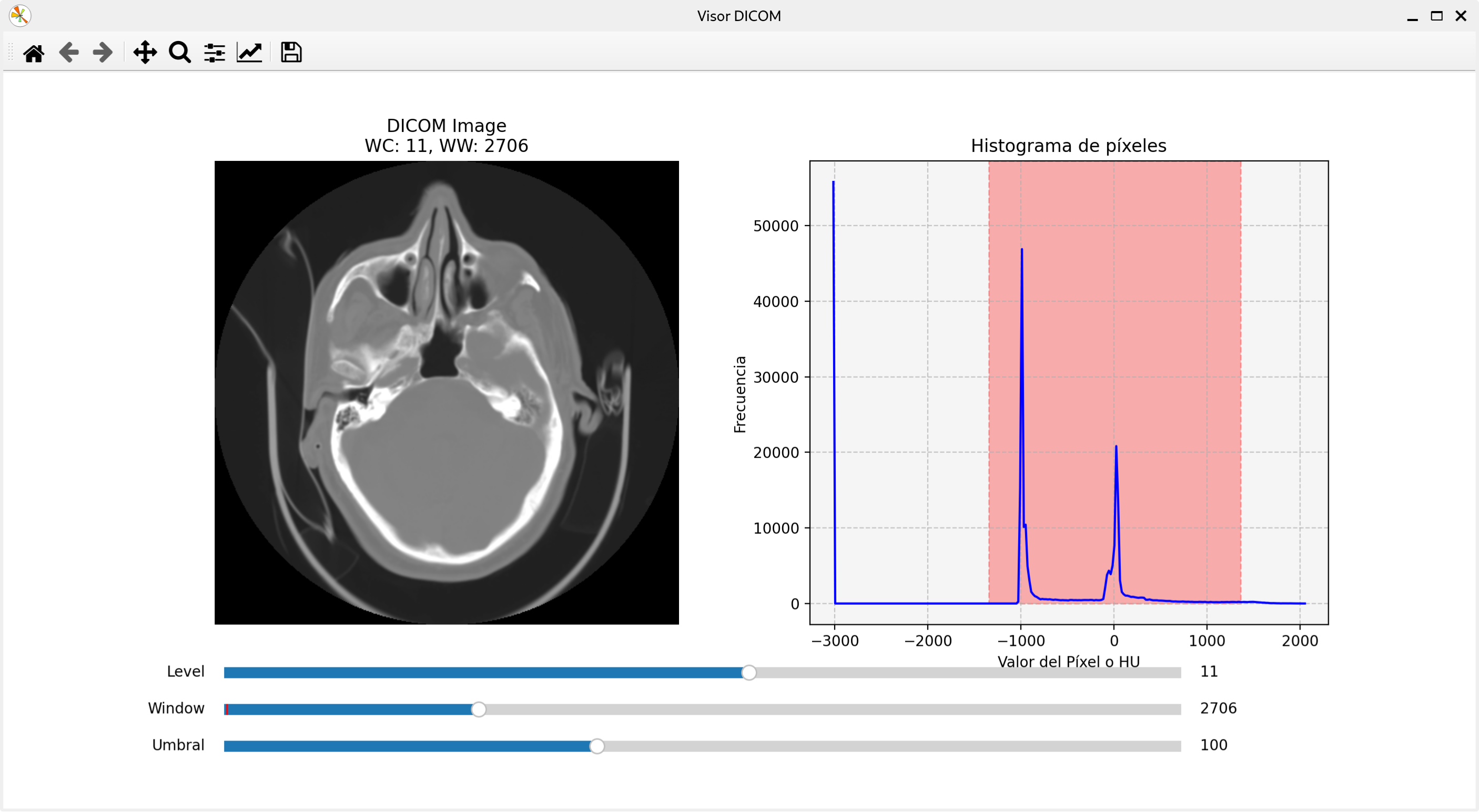
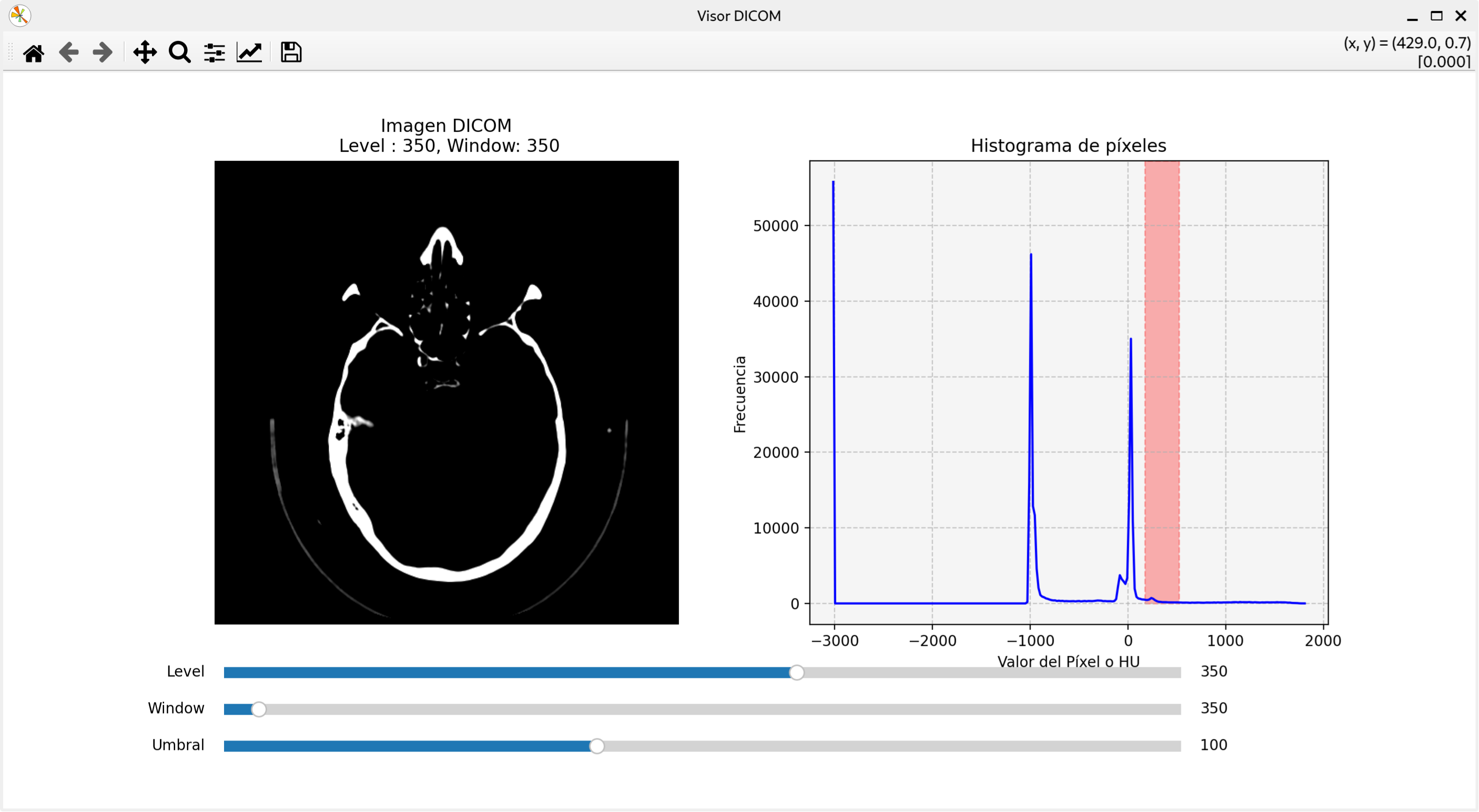
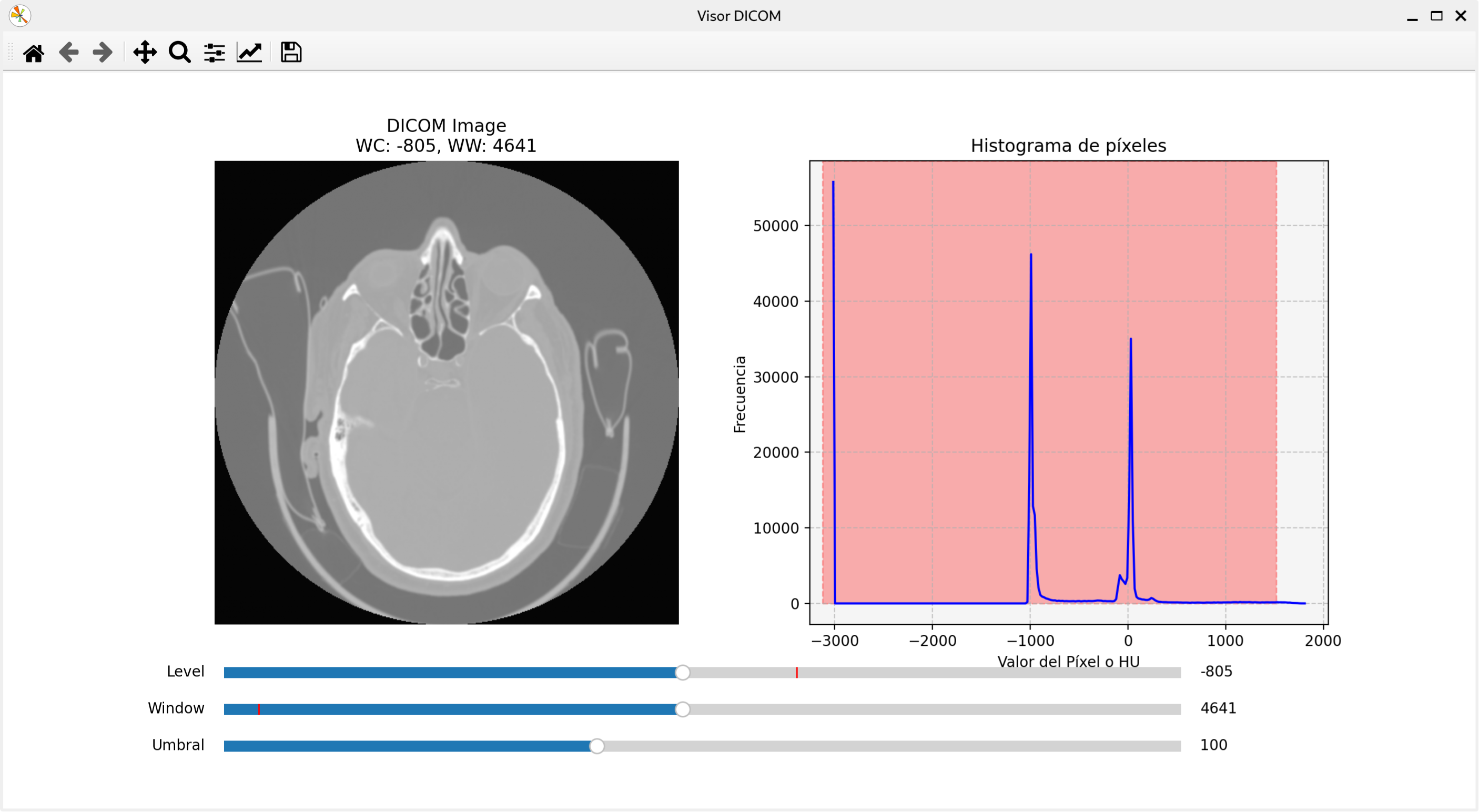
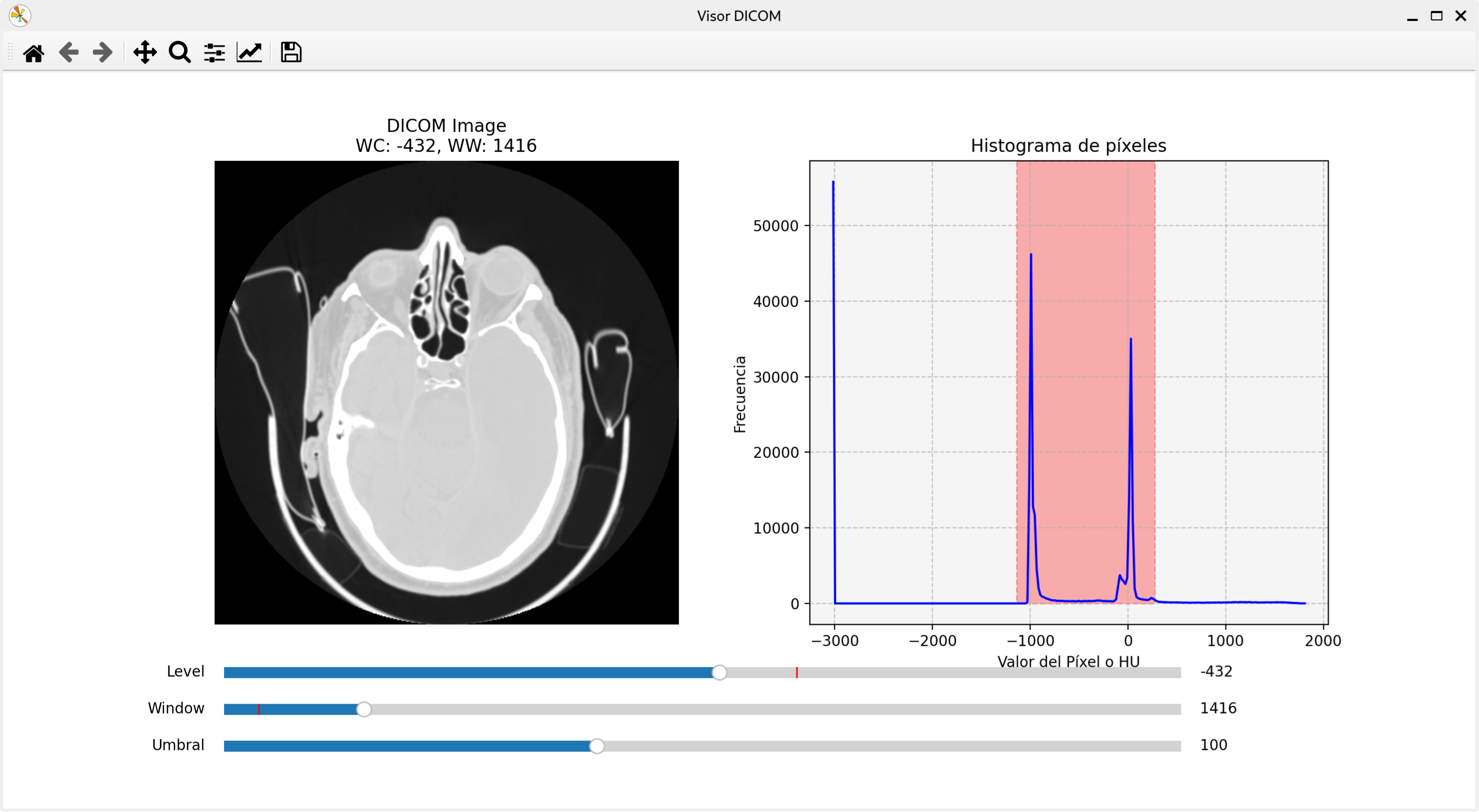
filedialog para abrir un archivo.Modificando los sliders, se pueden modificar las propiedades y aplicar la segmentación:
|
|
|
|
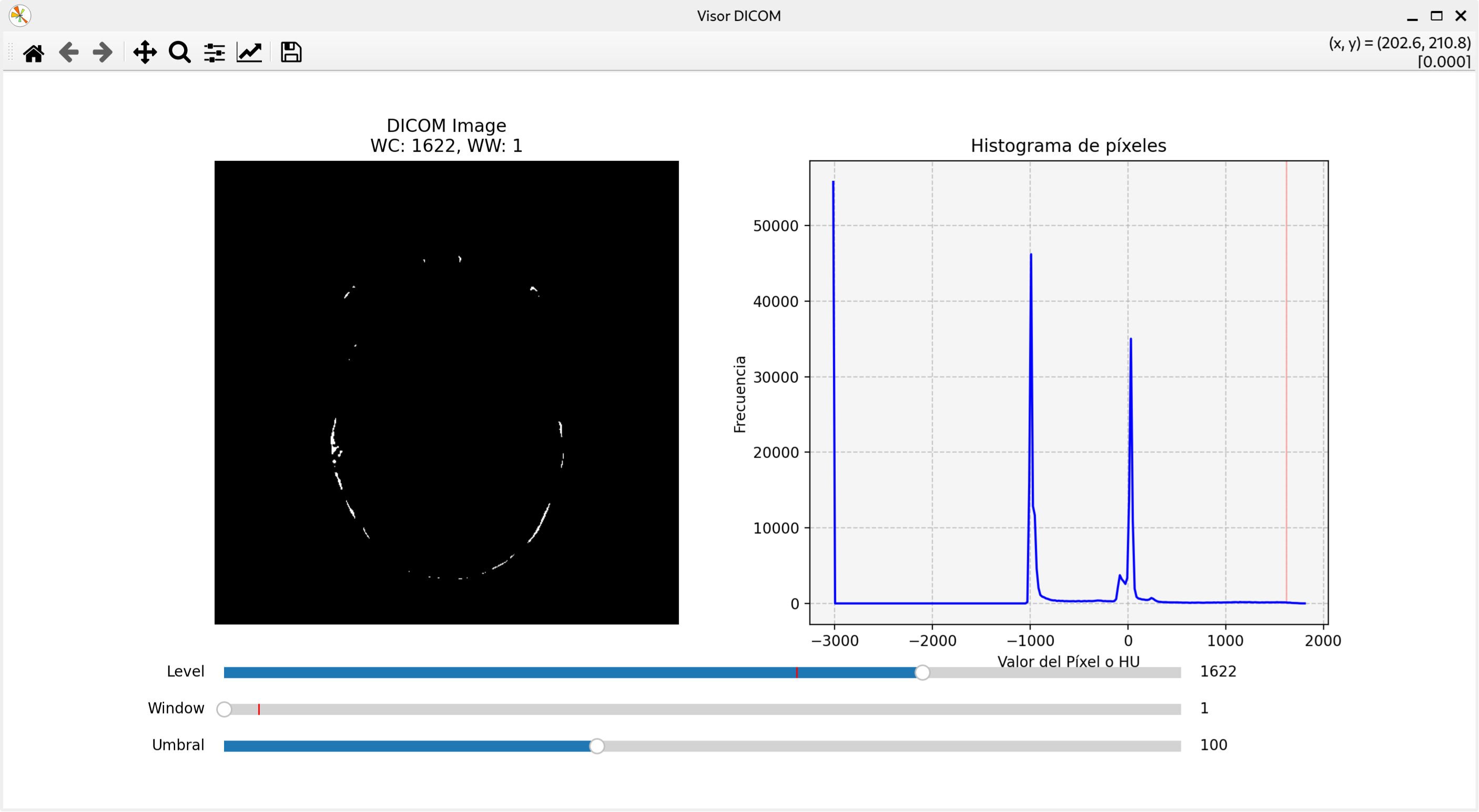
Como vemos a utilidad de los sliders de level y window es importante para posibilitar una correcta definición de diferentes regiones según su intensidad. Ello permitirá también saber el tipo de tejido al que miramos.
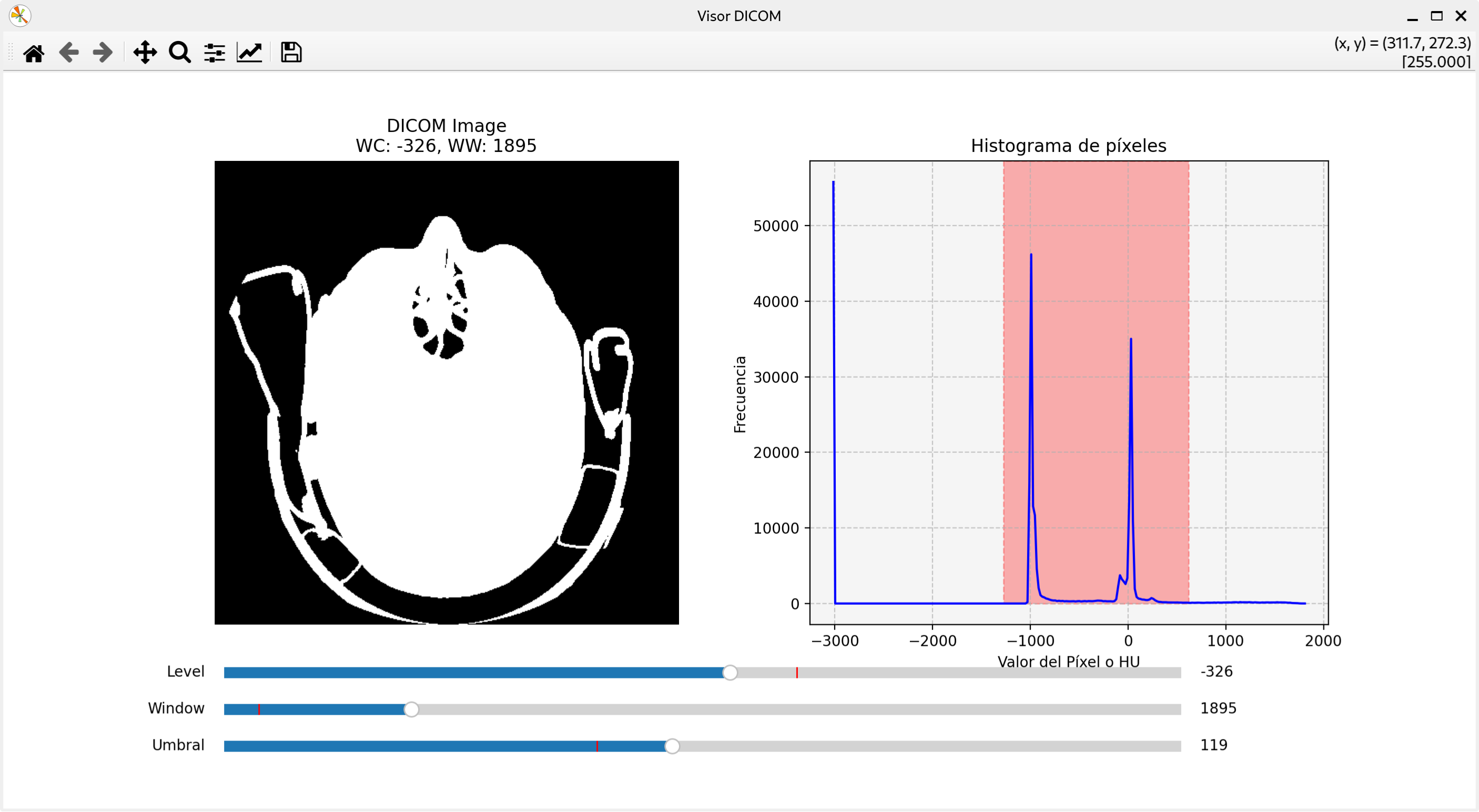
Comprobamos también la función del umbral:

|
|
Esta actividad ha permitido el acercamiento a la realidad del procesado de imágenes mediante el desarrollo e implementación de una interfaz gráfica de usuario para procesar imágenes médicas en el archivo estándar DICOM. Se han implementado técnicas de segmentación para las imágenes, así como una posibilidad para mejorar la visualización de las mismas, priorizando estructuras.
Las imágenes se han obtenido de (Adam, s.f.). El propio autor de la página especifica que no son suyas. El enlace que facilita (http://headctstudy.qure.ai/#dataset) está caído a fecha actual. Sin embargo, proporciona un enlace permalink en su página que aún funciona.
Adam, J. (s.f.). Dicom processing. Retrieved December 14, 2025, from https://jason-adam.github.io/dicom/
General Python FAQ - Python 3.14.0. (s.f). Python Documentation. https://docs.python.org/3/faq/general.html
Matplotlib documentation—Matplotlib 3.10.8 documentation. (s.f.). Retrieved December 14, 2025, from https://matplotlib.org/stable/index.html
Numpy documentation. (s.f.). Retrieved December 14, 2025, from https://numpy.org/doc/
Tkinter - Python interface to Tcl/Tk. (s.f). Python Documentation. https://docs.python.org/3/library/tkinter.html